近日,约翰·霍普金斯大学医学院Carol W. Greider团队在Science发表了题为“Human telomere length is chromosome end–specific and conserved across individuals”的文章,介绍了一种基于纳米孔测序技术的端粒分析方法——Telomere Profiling,可以单核苷酸分辨率测量细胞中每个端粒的长度。Carol W. Greider曾与Elizabeth Blackburn、Jack Szostak以”发现端粒和端粒酶是如何保护染色体的“这一研究成果,获得2009年诺贝尔生理学或医学奖。
研究团队利用这一新方法对 147 个个体的染色体端粒长度进行分析,发现端粒中位长度为 4.7kb,但不同染色体端粒长度差异极大,平均值相差超6kb。特别地,这种染色体末端特异性端粒长度差异具有个体保守性,在出生时就已确定,随年龄增长也得以保持。这一发现对于理解端粒生物学、衰老过程以及相关疾病的发生具有重要意义。综上,Telomere Profiling方法易于实施、结果精确并且成本较低,可广泛应用于科学研究和临床诊断,将使探索端粒生物学的全新领域成为可能。

文章发表在Science
主要研究内容:
1.纳米孔端粒分析准确且可重复报告端粒长度
为确定人类端粒是否在所有染色体上保持共同的长度分布,或者特定的染色体末端是否保持自己独特的长度分布,研究团队开发了一种富集、分析端粒的方法Telomere Profiling:首先使用生物素化的寡核苷酸(TeloTag)标记端粒末端;随后用链霉亲和素分离标记的端粒,并通过限制性内切酶酶切将其释放;最后通过牛津纳米孔技术(ONT)长读长测序方法对端粒进行测序。据悉,使用该方法检测每个样本的成本约为75美元。此外,研究团队还开发了新生物信息学分析流程来确定染色体末端特异性端粒长度。
接下来,研究团队通过对0岁至90岁人群的外周血单核细胞(PBMC)进行了端粒分析,并将其与Southern印迹法、FlowFISH检测的结果进行对比。结果显示,经不同方法所检测的端粒长度高度一致,表明Telomere Profiling方法具有高度准确性及优异可重复性。此外,研究团队还通过检测7个样本的端粒长度来检测实验室间的差异性,确认了该方法的广泛适用性和可靠性。

图1. 纳米孔技术进行端粒分析是准确和精确的
2.端粒长度随年龄增长而发生变化
已知端粒长度随着年龄的增长而缩短,但先前方法无法在核苷酸分辨率上测量端粒长度。为检测端粒长度动态范围,研究团队使用Telomere Profiling对11个个体(0-84岁)的DNA样本进行分析,并根据端粒长度进行排序;通过Southern印迹法对相同的DNA进行测量,并将其作为验证。结果显示,Telomere Profiling预测了端粒长度的等级顺序,捕获了Southern印迹的动态范围,并测量端粒随年龄增长而缩短的情况,这对于理解衰老过程中的生物学变化至关重要。此外,Telomere Profiling还确定了端粒长度的第1、第10和第50百分位数。
研究团队还将该方法与FlowFISH进行了比较,使用先前诊断为短端粒综合征的特发性肺纤维化(IPF)患者的5μg存档DNA样本进行分析。结果显示,大多数IPF样本的整体端粒长度与FlowFISH测量结果相似,表明Telomere Profiling能够检测出患病个体,有望助力临床诊断和治疗。
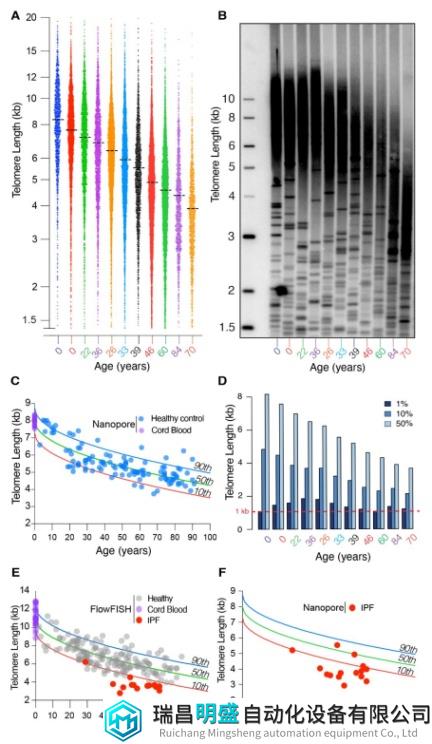
图2. 纳米孔端粒分析检测端粒长度随年龄的动态变化
3.人类端粒具有染色体末端特异性长度和单倍型特异性长度差异
为确定人类是否具有染色体末端特异性端粒长度,研究团队分析了来自二倍体HG002细胞系的端粒,从HG002细胞系中分离DNA,并对端粒进行测序,将平均总长度为16.4 kb的reads映射到HG002参考基因组中,共有77个染色体末端通过质量筛选。结果显示,每条染色体的末端表现出不同的端粒长度分布;端粒中位长度为 4.7kb,有66个端粒的长度分布与均值有显著差异,平均长度差异超过6kb。除染色体末端特异性长度外,一些端粒在母系和父系单倍型之间也存在显著差异。上述结果表明,人类端粒具有染色体末端特异性长度分布。
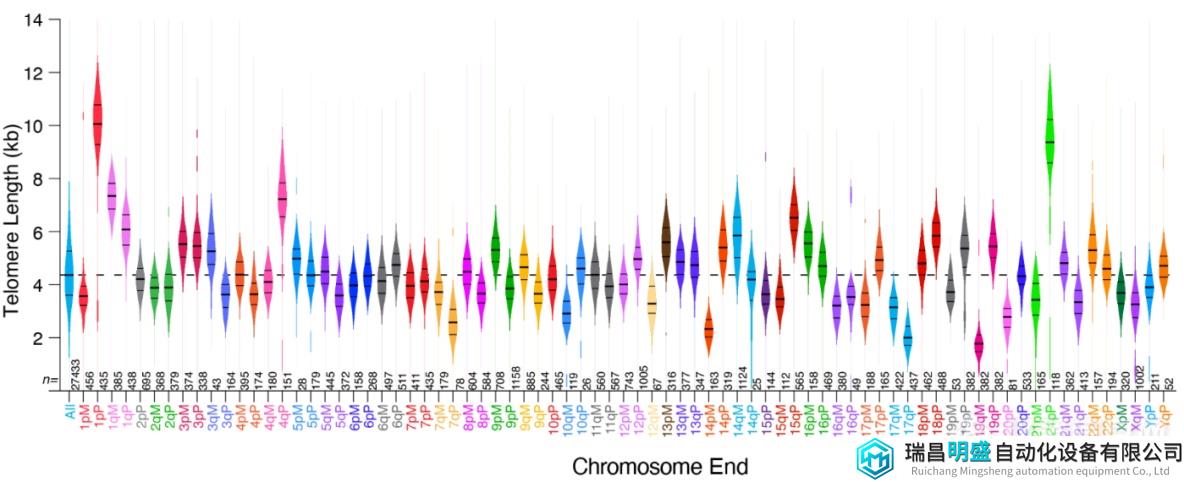
图3. 染色体末端特异性端粒长度
4.染色体特异性端粒长度在个体间是保守的
研究团队将150个个体的端粒序列与最近发布的泛基因组中的亚端粒序列进行了比对,将300个单倍体基因组的全基因组序列与3个高质量单倍体参考T2T基因组CHM13、HG002母基因组和HG002父基因组的序列进行比对,以确定每个参考基因组中相同亚端粒的可重复性。在所有reads中,87%在泛基因组和CHM13中定位到相同的染色体末端,90%在泛基因组和HG002母系中定位到相同的染色体末端,88%在泛基因组和HG002父系中定位到相同的染色体末端。这些数据表明,经Telomere Profiling检测的reads均可映射到一个特定的泛基因组染色体图谱,具有很高的可信度。
研究团队建立了相对平均端粒长度,分析了147个个体PBMC样本每个染色体末端端粒长度,并根据端粒的相对长度对染色体末端进行排序。结果显示,17p、20q和12p往往是群体中最短的端粒,而4q、12q和3p往往是最长的端粒。因此,虽然在单个个体中可以看到端粒长度的单倍型特异性差异,但在整个群体中,某些染色体末端更有可能比总体平均值短,而其他染色体末端更有可能比总体平均值长。研究团队还分析了不同年龄段的样本,在婴儿脐带血样本发现了同样的端粒长度差异,说明随着年龄增长,端粒长度普遍缩短,但长度差异保持不变。这些结果表明,个体端粒长度的差异是在出生时就已存在,且在不同个体间是保守的。
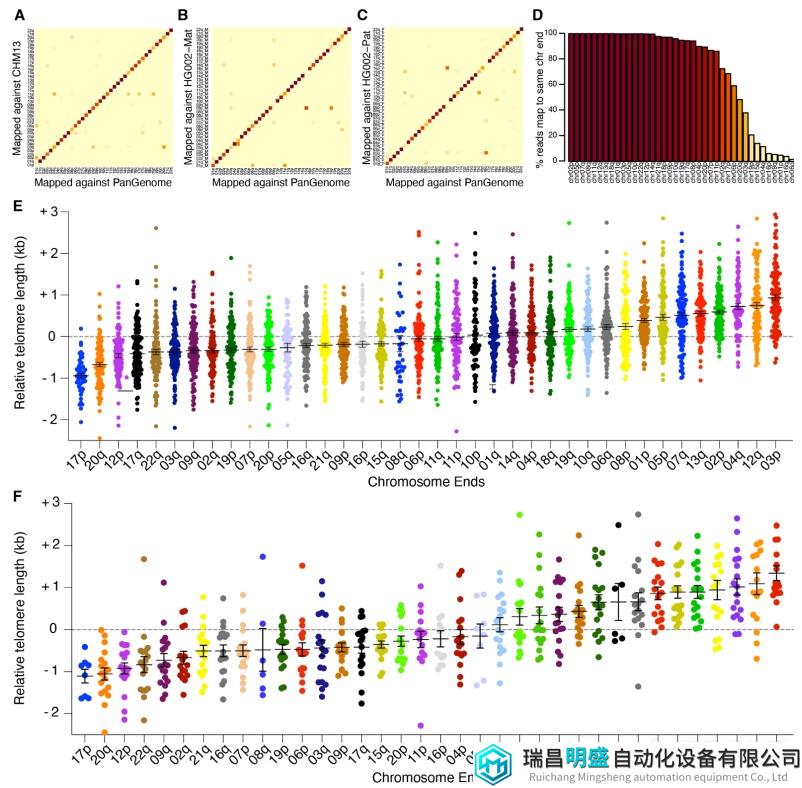
图4. 染色体特异性端粒长度在人群中是保守的
综上所述,研究团队开发了一种简单通用的端粒富集分析方法Telomere Profiling,并跨越了广泛的年龄范围,基于该方法发现了染色体特异性和单倍型特异性的端粒长度分布,其中一些端粒长度之间存在显著差异,拓展了端粒长度的临床意义。综上,Telomere Profiling将帮助人们更深入地了解端粒生物学,并有望推动相关领域发展,从而有望找到治疗疾病的新方法。
研究团队指出:“Telomere Profiling可使精确的端粒长度研究广泛应用于实验室、临床和药物发现工作中。因此,在未来的研究中,应该加强不同人群的检测,以证实某些染色体末端是否始终是最短的还是最长的。”
原文链接:
K. Karimian et al., Human telomere length is chromosome end–specific and conserved across individuals. Science (2024). https://www.science.org/doi/10.1126/science.ado0431






